library(canregtools)
library(dplyr)
files <- list.files("~/website/slides/outputs", full.names = TRUE)
file <- files[1]
data <- read_canreg(file)
class(data)[1] "canreg" "list" [1] "areacode" "FBcases" "SWcases" "POP" Report cancer registration data using a developed R package canregtools
陈琼
河南省癌症中心/河南省肿瘤医院
Monday Oct 28, 2024(南京)
内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!
肿瘤登记是对肿瘤流行情况、趋势变化和影响因素进行长期、连续、动态的系统性监测,是制定癌症防治策略、开展综合研究、评价防控效果的重要基础性工作。
内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!

Canregtools 是一个旨在使肿瘤登记数据的分析、可视化和报告更加顺畅和高效的 R 语言包。它通过一系列R函数实现数据读取、数据处理、统计计算、可视化和报告等功能,全面支持 R 环境中的工作流程。
Canregtools 是一个为不同级别肿瘤登记处设计的工具。它支持批量处理来自多个登记处的数据,允许用户根据自定义条件筛选数据,并根据登记处属性重新格式化或合并肿瘤登记数据。
我们定义了一组不同的类,通过泛函数对于不同的类执行不同的函数功能。
create_asr, create_quality, create_age_rate, create_sheet, cr_select, cr_merge, reframe_fbswicd
内部一致性检查是进行数据分析之前的关键步骤。我们需要识别并处理任何不可能或不太可能的变量组合,以确保数据的有效性。


目前canregtools包还处在功能不断完善阶段,还没有往CRAN正式提交,目前可以通过github安装或者本地文件安装。
从github安装
# install the remotes package if doesn't installed
install.packages("remotes)
library(remotes)
install_github("gigu003/canregtools")从本地编译文件安装
内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!
Canregtools可以读取NCC call for data 格式原始数据,一个包含三个名为FB、SW和POP工作表的Excel文件,分别存储发病数据、死亡数据和人口数据。


“canreg” 是一个包含四个元素的列表,这些元素被命名为 ‘areacode’、‘FBcases’、‘SWcases’ 和 ‘POP’,它们是从原始数据的 “FB”、“SW” 和 “POP” 工作表中读取的。
[1] "areacode" "FBcases" "SWcases" "POP" [1] "410102"# A tibble: 6 × 20
registr sex birthda addcode trib occu marri inciden topo morp
<chr> <chr> <date> <chr> <chr> <chr> <chr> <date> <chr> <chr>
1 21410500166… 1 1975-04-17 410102… 01 31 2 2021-10-26 C15.9 8010
2 21410802159… 2 1991-01-16 410102… 01 14 2 2021-10-22 C53.8 8140
3 21410102172… 2 1962-10-15 410102… 01 00 2 2021-01-19 C53.0 8070
4 21411202123… 2 1951-03-09 410102… 01 49 2 2021-01-30 C50.9 8000
5 21411624137… 1 1955-07-15 410102… 01 61 2 2021-05-13 C22.0 8000
6 23411002105… 1 1939-12-15 410102… 01 28 5 2021-12-22 C34.1 8140
# ℹ 10 more variables: beha <chr>, grad <chr>, basi <chr>, icd10 <chr>,
# autoicd10 <chr>, lastcontact <dttm>, status <chr>, caus <chr>,
# deathda <date>, deadplace <chr>“canreg” 是一个包含四个元素的列表,这些元素被命名为 ‘areacode’、‘FBcases’、‘SWcases’ 和 ‘POP’,它们是从原始数据的 “FB”、“SW” 和 “POP” 工作表中读取的。
[1] "areacode" "FBcases" "SWcases" "POP" # A tibble: 6 × 19
registr sex birthda trib occu marri inciden topo morp beha grad
<chr> <chr> <date> <chr> <chr> <chr> <date> <chr> <chr> <chr> <chr>
1 1741010… 2 1921-02-18 01 39 2 2014-10-21 C44.5 8010 3 9
2 1841010… 1 1935-05-15 01 80 2 2018-07-14 C34.9 8010 3 9
3 1841010… 2 1941-02-16 01 29 2 2015-04-02 C34.9 8000 3 9
4 1841010… 1 1952-07-06 01 90 2 2018-04-17 C61.9 8140 3 3
5 1841010… 1 1947-08-21 01 85 2 2017-11-13 C16.3 8140 3 9
6 1841010… 1 1941-08-16 01 29 2 2015-12-23 C73.9 8050 3 9
# ℹ 8 more variables: basi <chr>, icd10 <chr>, autoicd10 <chr>,
# lastcontact <dttm>, status <chr>, caus <chr>, deathda <date>,
# deadplace <chr>“canreg” 是一个包含四个元素的列表,这些元素被命名为 ‘areacode’、‘FBcases’、‘SWcases’ 和 ‘POP’,它们是从原始数据的 “FB”、“SW” 和 “POP” 工作表中读取的。
count_canreg()函数可以把’canreg’类数据转换为’fbswicd’类数据,’fbswicd’类数据是一个按照指定分类方法和格式转换的汇总数据,当原始数据量很大时,可以节省存储空间和运行效率。
[1] "fbswicd" "list" [1] "areacode" "fbswicd" "sitemorp" "pop" year sex agegrp cancer fbs sws mv ub sub m8000 dco
<int> <int> <fctr> <int> <int> <int> <int> <int> <int> <int> <int>
1: 2021 1 0 岁 60 3 0 2 1 1 1 0
2: 2021 1 0 岁 61 3 0 2 1 1 1 0 year sex cancer site morp
<int> <int> <int> <list> <list>
1: 2021 2 115 <data.frame[3x2]> <data.frame[9x2]>
2: 2021 2 114 <data.frame[8x2]> <data.frame[18x2]> year sex agegrp rks
<int> <int> <fctr> <int>
1: 2021 1 0 岁 3850
2: 2021 1 1-4 岁 14405summary 函数可以快速提取’canreg’类数据摘要,比如发病率、死亡率、死亡发病比、数据格式检查情况、人口数据检查情况、基本变量符合情况等,从而为多登记处的筛选提供数据。
[1] "summary" "list" [1] "areacode" "rks" "fbs" "inci"
[5] "sws" "mort" "mi" "mv"
[9] "dco" "rks_year" "inci_vars" "miss_r_vars_inci"
[13] "mort_vars" "miss_r_vars_mort"[1] 0.38[1] 373.15[1] 142create_asr 函数能够根据提供的标准人口计算年龄标准化率、截缩率和累积率,它还可以估计标化率的方差和95%置信区间。
# A tibble: 5 × 2
Vars Description
<chr> <chr>
1 cn64 Standard population in Chinese in 1964
2 cn82 Standard population in Chinese in 1982
3 cn2000 Standard population in Chinese in 2000
4 wld85 Segi's world standard population
5 wld2000 World standard population in 2000 # calculate asr using the create_asr() function
create_asr(fbsw, event = fbs, year, sex, cancer, std = c("cn2000", "wld85"))# A tibble: 56 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <int> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 1069 327. 266. 267. 436.
2 2021 1 61 1050 321. 262. 263. 429.
3 2021 1 101 18 5.50 4.68 4.84 10.4
4 2021 1 102 5 1.53 1.23 1.45 2.63
5 2021 1 103 47 14.4 10.8 10.3 12.0
6 2021 1 104 65 19.9 15.6 16.0 21.9
7 2021 1 105 87 26.6 21.0 21.7 32.9
8 2021 1 106 84 25.7 20.7 21.7 39.4
9 2021 1 107 21 6.41 5.12 5.57 9.64
10 2021 1 108 28 8.55 6.82 7.14 10.1
# ℹ 46 more rows
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>一些后处理函数如drop_total, drop_others, and add_labels 可以对生成的数据进行癌种筛选、添加标签等操作。
create_asr(fbsw, event = fbs, year, sex, cancer) |>
drop_total() |> drop_others() |>
add_labels(lang = "en", label_type = "abbr")# A tibble: 48 × 13
year sex cancer site icd10 no_cases cr asr_cn2000 asr_wld85
<int> <fct> <int> <fct> <fct> <int> <dbl> <dbl> <dbl>
1 2021 Male 101 Oral Cavity & P… C00-… 18 5.50 4.68 4.84
2 2021 Male 102 Nasopharynx C11 5 1.53 1.23 1.45
3 2021 Male 103 Esophagus C15 47 14.4 10.8 10.3
4 2021 Male 104 Stomach C16 65 19.9 15.6 16.0
5 2021 Male 105 Colorectum C18-… 87 26.6 21.0 21.7
6 2021 Male 106 Liver C22 84 25.7 20.7 21.7
7 2021 Male 107 Gallbladder C23-… 21 6.41 5.12 5.57
8 2021 Male 108 Pancreas C25 28 8.55 6.82 7.14
9 2021 Male 109 Larynx C32 15 4.58 3.72 3.97
10 2021 Male 110 Lung C33-… 230 70.2 57.1 59.1
# ℹ 38 more rows
# ℹ 4 more variables: truncr_cn2000 <dbl>, truncr_wld85 <dbl>, cumur <dbl>,
# prop <dbl>create_quality函数可以基于“canreg”类数据或“fbswicd”类数据计算质量指标,包括癌症病例数、粗发病率、死亡率、死亡发病比、形态学诊断的病例比例、DCO(死亡证明诊断病例比例)、UB%等。
# calculate quality indicators based on 'canreg' data
create_quality(data, year, sex, cancer) |> filter(!cancer == 0) |>
add_labels(lang = "en")# A tibble: 56 × 16
year sex cancer site icd10 rks fbs fbl sws swl mi mv
<int> <fct> <int> <fct> <fct> <int> <int> <dbl> <int> <dbl> <dbl> <dbl>
1 2021 Male 60 All Ca… ALL 327403 1069 327. 559 171. 0.52 76.8
2 2021 Male 61 All Ca… ALLb… 327403 1050 321. 553 169. 0.53 76.6
3 2021 Male 101 Oral C… C00-… 327403 18 5.5 10 3.05 0.56 55.6
4 2021 Male 102 Nasoph… C11 327403 5 1.53 4 1.22 0.8 40
5 2021 Male 103 Esopha… C15 327403 47 14.4 35 10.7 0.74 87.2
6 2021 Male 104 Stomach C16 327403 65 19.8 55 16.8 0.85 73.8
7 2021 Male 105 Conlon… C18-… 327403 87 26.6 55 16.8 0.63 77.0
8 2021 Male 106 Liver C22 327403 84 25.7 77 23.5 0.92 66.7
9 2021 Male 107 Gallbl… C23-… 327403 21 6.41 16 4.89 0.76 85.7
10 2021 Male 108 Pancre… C25 327403 28 8.55 28 8.55 1 50
# ℹ 46 more rows
# ℹ 4 more variables: dco <dbl>, ub <dbl>, sub <dbl>, m8000 <dbl># calculate quality indicators based on 'fbswicd' data
create_quality(fbsw, year, sex) |>
add_labels(lang = "en")# A tibble: 2 × 16
year sex cancer site icd10 rks fbs fbl sws swl mi mv
<int> <fct> <dbl> <fct> <fct> <int> <int> <dbl> <int> <dbl> <dbl> <dbl>
1 2021 Male 60 All Canc… ALL 327403 1069 327. 559 171. 0.52 76.8
2 2021 Female 60 All Canc… ALL 355707 1415 398. 405 114. 0.29 84.8
# ℹ 4 more variables: dco <dbl>, ub <dbl>, sub <dbl>, m8000 <dbl># A tibble: 28 × 16
year sex cancer site icd10 rks fbs fbl sws swl mi mv
<dbl> <fct> <int> <fct> <fct> <int> <int> <dbl> <int> <dbl> <dbl> <dbl>
1 9000 Total 60 All Ca… ALL 683110 2484 364. 964 141. 0.39 81.4
2 9000 Total 61 All Ca… ALLb… 683110 2451 359. 950 139. 0.39 81.2
3 9000 Total 101 Oral C… C00-… 683110 26 3.81 13 1.9 0.5 57.7
4 9000 Total 102 Nasoph… C11 683110 7 1.02 5 0.73 0.71 57.1
5 9000 Total 103 Esopha… C15 683110 66 9.66 46 6.73 0.7 86.4
6 9000 Total 104 Stomach C16 683110 101 14.8 79 11.6 0.78 71.3
7 9000 Total 105 Conlon… C18-… 683110 181 26.5 103 15.1 0.57 81.2
8 9000 Total 106 Liver C22 683110 125 18.3 100 14.6 0.8 68
9 9000 Total 107 Gallbl… C23-… 683110 44 6.44 31 4.54 0.7 86.4
10 9000 Total 108 Pancre… C25 683110 46 6.73 57 8.34 1.24 43.5
# ℹ 18 more rows
# ℹ 4 more variables: dco <dbl>, ub <dbl>, sub <dbl>, m8000 <dbl>create_age_rate 函数可以基于“canreg”类数据或“fbswicd”类数据计算年龄别率,并输出为不同的格式(长数据或宽数据)。
# calculate age specific rate from 'canreg' data.
create_age_rate(data, year, sex, cancer, format = "long") |>
filter(!cancer == 0) |>
arrange(year, sex, cancer, agegrp)# A tibble: 1,064 × 6
year sex cancer agegrp cases rate
<int> <int> <int> <fct> <int> <dbl>
1 2021 1 60 0 岁 3 77.9
2 2021 1 60 1-4 岁 3 20.8
3 2021 1 60 5-9 岁 5 32.9
4 2021 1 60 10-14 岁 0 0
5 2021 1 60 15-19 岁 2 10.7
6 2021 1 60 20-24 岁 5 18.0
7 2021 1 60 25-29 岁 14 38.7
8 2021 1 60 30-34 岁 38 130.
9 2021 1 60 35-39 岁 36 121.
10 2021 1 60 40-44 岁 60 196.
# ℹ 1,054 more rows# calculate age specific rate from 'fbswicd' data.
create_age_rate(fbsw, year, sex, cancer, format = "wide") |>
filter(!cancer == 0) |>
add_labels(lang = "en")# A tibble: 56 × 45
year sex cancer site icd10 f0 f1 f2 f3 f4 f5 f6
<int> <fct> <int> <fct> <fct> <int> <int> <int> <int> <int> <int> <int>
1 2021 Male 60 All Cance… ALL 1069 3 3 5 0 2 5
2 2021 Male 61 All Cance… ALLb… 1050 3 3 5 0 2 5
3 2021 Male 101 Oral Cavi… C00-… 18 0 0 0 0 0 0
4 2021 Male 102 Nasophary… C11 5 0 0 0 0 0 0
5 2021 Male 103 Esophagus C15 47 0 0 0 0 0 0
6 2021 Male 104 Stomach C16 65 0 0 0 0 0 0
7 2021 Male 105 Conlon, R… C18-… 87 0 0 0 0 0 0
8 2021 Male 106 Liver C22 84 0 1 0 0 0 0
9 2021 Male 107 Gallbladd… C23-… 21 0 0 0 0 0 0
10 2021 Male 108 Pancreas C25 28 0 0 0 0 0 0
# ℹ 46 more rows
# ℹ 33 more variables: f7 <int>, f8 <int>, f9 <int>, f10 <int>, f11 <int>,
# f12 <int>, f13 <int>, f14 <int>, f15 <int>, f16 <int>, f17 <int>,
# f18 <int>, f19 <int>, r0 <dbl>, r1 <dbl>, r2 <dbl>, r3 <dbl>, r4 <dbl>,
# r5 <dbl>, r6 <dbl>, r7 <dbl>, r8 <dbl>, r9 <dbl>, r10 <dbl>, r11 <dbl>,
# r12 <dbl>, r13 <dbl>, r14 <dbl>, r15 <dbl>, r16 <dbl>, r17 <dbl>,
# r18 <dbl>, r19 <dbl>内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!
read_canreg 函数可以读取多个原始数据并将其转换为’canregs’类数据,其实’canregs’ 类数据是一个包含多个元素的列表,每个元素是一个’canreg’类数据。
summary 函数可以快速地计算’canregs’类数据的摘要信息,并通过cr_select函数对指定的条件进行筛选。
[1] "410102" "410103" "410104" "410105" "410123" "410185" "410224" "410302"
[9] "410303" "410304"[1] "410302" "410303" "410304"[1] "410102" "410103" "410104" "410105" "410302" "410303" "410304"[1] "410102" "410303"cr_select 函数可以通过指定一个或多个条件对’carengs’, ‘fbswicds’, ‘asrs’, and, ’summaries’类数据进行筛选和过滤,从而满足通过一定条件对登记处数据筛选的功能。
count_canreg 函数可以转换 ‘canregs’ 类数据为’fbswicds’类数据, ’fbswicds’类数据是一个列表,列表的每个元素是一个’fbswicd’类数据,其可以作为create_asr, create_quality, create_sheet, 等函数的输入数据。
create_asr 函数可以基于’canregs’或着’fbswicds’类数据计算年龄标化率.
[1] "asrs" "list"$`410102`
# A tibble: 2 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 1069 327. 266. 267. 436.
2 2021 2 60 1415 398. 319. 299. 565.
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>
$`410103`
# A tibble: 2 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 1190 362. 242. 237. 380.
2 2021 2 60 1469 409. 289. 272. 539.
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>
$`410104`
# A tibble: 2 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 720 316. 244. 239. 398.
2 2021 2 60 862 341. 272. 258. 500.
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>
$`410105`
# A tibble: 2 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 1816 392. 286. 273. 429.
2 2021 2 60 2334 461. 344. 319. 610.
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>
$`410302`
# A tibble: 2 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 289 348. 225. 222. 345.
2 2021 2 60 272 315. 188. 182. 343.
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>
$`410303`
# A tibble: 2 × 11
year sex cancer no_cases cr asr_cn2000 asr_wld85 truncr_cn2000
<int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 625 383. 278. 310. 280.
2 2021 2 60 596 369. 230. 222. 354.
# ℹ 3 more variables: truncr_wld85 <dbl>, cumur <dbl>, prop <dbl>cr_merge 函数可以合并 ‘carengs’, ‘fbswicds’, ‘asrs’, 和 ‘qualities’类数据为 ’canreg’, ‘fbswicd’, ‘asr’, 和 ‘quality’ 类数据.
# A tibble: 6 × 8
areacode year sex cancer no_cases cr asr_cn2000 asr_wld85
<int> <int> <int> <dbl> <int> <dbl> <dbl> <dbl>
1 410102 2021 1 60 1069 327. 266. 267.
2 410102 2021 2 60 1415 398. 319. 299.
3 410103 2021 1 60 1190 362. 242. 237.
4 410103 2021 2 60 1469 409. 289. 272.
5 410104 2021 1 60 720 316. 244. 239.
6 410104 2021 2 60 862 341. 272. 258. [1] "areacode" "year" "sex" "cancer"
[5] "no_cases" "cr" "asr_cn2000" "asr_wld85"
[9] "truncr_cn2000" "truncr_wld85" "cumur" "prop" $`410102`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 327403 1069 327. 559 171. 0.52 76.8 1.5 1.78
2 2021 2 60 355707 1415 398. 405 114. 0.29 84.8 0.85 1.98
# ℹ 2 more variables: sub <dbl>, m8000 <dbl>
$`410103`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 328415 1190 362. 672 205. 0.56 73.9 0.67 2.18
2 2021 2 60 358941 1469 409. 408 114. 0.28 77.9 1.09 2.11
# ℹ 2 more variables: sub <dbl>, m8000 <dbl>
$`410104`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 227511 720 316. 369 162. 0.51 82.2 0.56 1.81
2 2021 2 60 253077 862 341. 252 99.6 0.29 82.2 0.35 1.28
# ℹ 2 more variables: sub <dbl>, m8000 <dbl>
$`410105`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 462738 1816 392. 792 171. 0.44 77.8 0.72 1.6
2 2021 2 60 506456 2334 461. 553 109. 0.24 80.9 0.21 1.63
# ℹ 2 more variables: sub <dbl>, m8000 <dbl>
$`410302`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub sub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 83010 289 348. 153 184. 0.53 69.9 0 1.04 65.7
2 2021 2 60 86359 272 315. 103 119. 0.38 75.4 0.74 0.74 72.1
# ℹ 1 more variable: m8000 <dbl>
$`410303`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 163005 625 383. 412 253. 0.66 76.8 1.12 0.96
2 2021 2 60 161724 596 369. 252 156. 0.42 84.4 1.51 2.52
# ℹ 2 more variables: sub <dbl>, m8000 <dbl>
$`410304`
# A tibble: 2 × 14
year sex cancer rks fbs fbl sws swl mi mv dco ub
<int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2021 1 60 99735 427 428. 232 233. 0.54 71.4 2.81 2.81
2 2021 2 60 100963 353 350. 147 146. 0.42 79.3 2.55 1.13
# ℹ 2 more variables: sub <dbl>, m8000 <dbl>
attr(,"class")
[1] "qualities" "list" # A tibble: 6 × 12
areacode year sex cancer rks fbs fbl sws swl mi mv dco
<int> <int> <int> <dbl> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
1 410102 2021 1 60 327403 1069 327. 559 171. 0.52 76.8 1.5
2 410102 2021 2 60 355707 1415 398. 405 114. 0.29 84.8 0.85
3 410103 2021 1 60 328415 1190 362. 672 205. 0.56 73.9 0.67
4 410103 2021 2 60 358941 1469 409. 408 114. 0.28 77.9 1.09
5 410104 2021 1 60 227511 720 316. 369 162. 0.51 82.2 0.56
6 410104 2021 2 60 253077 862 341. 252 99.6 0.29 82.2 0.35 [1] "areacode" "year" "sex" "cancer" "rks" "fbs"
[7] "fbl" "sws" "swl" "mi" "mv" "dco"
[13] "ub" "sub" "m8000" 内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!
tidy_areacode 函数可以显示肿瘤登记处属性,并根据属性值进行分组。
[1] "areacode" "registry" "province" "city" "area_type" "region" 可以使用write_registry, or write_area_type函数修改登记处属性。
reframe_fbswicd函数可以根据登记处属性如’area_type’, ‘registry’, ‘province’把’fbswicds’转换为’fbswicd’。
# A tibble: 4 × 8
areacode year sex cancer no_cases cr asr_cn2000 asr_wld85
<int> <dbl> <int> <dbl> <int> <dbl> <dbl> <dbl>
1 910000 9000 1 60 6136 363. 253. 248.
2 910000 9000 2 60 7301 400. 288. 270.
3 920000 9000 1 60 2676 233. 172. 168.
4 920000 9000 2 60 2817 254. 186. 176. [1] "areacode" "year" "sex" "cancer"
[5] "no_cases" "cr" "asr_cn2000" "asr_wld85"
[9] "truncr_cn2000" "truncr_wld85" "cumur" "prop" 内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!


library(dplyr)
asr1 <- create_asr(fbsw,year,sex,cancer,event = fbs) |> mutate(type="incidence")
asr2 <- create_asr(fbsw,year,sex,cancer,event = sws) |> mutate(type="mortality")
asr <- bind_rows(asr1, asr2) |> drop_others() |> drop_total() |>
add_labels(label_type = "abbr",lang = "en")
draw_barchart(asr, plot_var =cr, cate_var = site,group_var = type,
side_label = c("Male","Female"))
[1] "year" "sex" "cancer" "site" "icd10" "agegrp" "cases" "rate" 
内容
canregtools 初衷
Canregtools 功能概述
单个肿瘤登记处
批量处理多登记处数据
数据合并和转换
肿瘤登记数据的可视化
自动化报告
谢谢关注!
肿瘤登记处的主要目标之一就是产生肿瘤发病、死亡、生存相关的统计指标, 并通过一定的渠道发布数据,促进数据的共享和利用(专业人员和大众),从而促进癌症防控政策的产生。
flowchart TD A(数据来源单位) --> B(县区级登记处) B --> C(市级登记处) C --> D(省级质量控制) D --> B B --> A D --> C D --> F(综合评估) F --> G(年报撰写及发布)
把统计分析数据转换为文字的过程。
flowchart LR
A(统计分析) --> B(制作表格)
B --> C(绘制统计图)
C --> D(专家撰写)
D --> E(初稿)
E --> F(审核定版)
年报特点
自动化?
基于现有年报的特点,以及随着可重复性报告技术的成熟,可以实现自动化撰写的需求?
思路
利用Bookdown包把统计结果进行展现,包括文字描述和可视化展现(各种统计图表)
Bookdown程序由按照章节区分的Rmd文件组成,每个Rmd文件可以输出相应章节的文字描述,从而形成最终的文本。
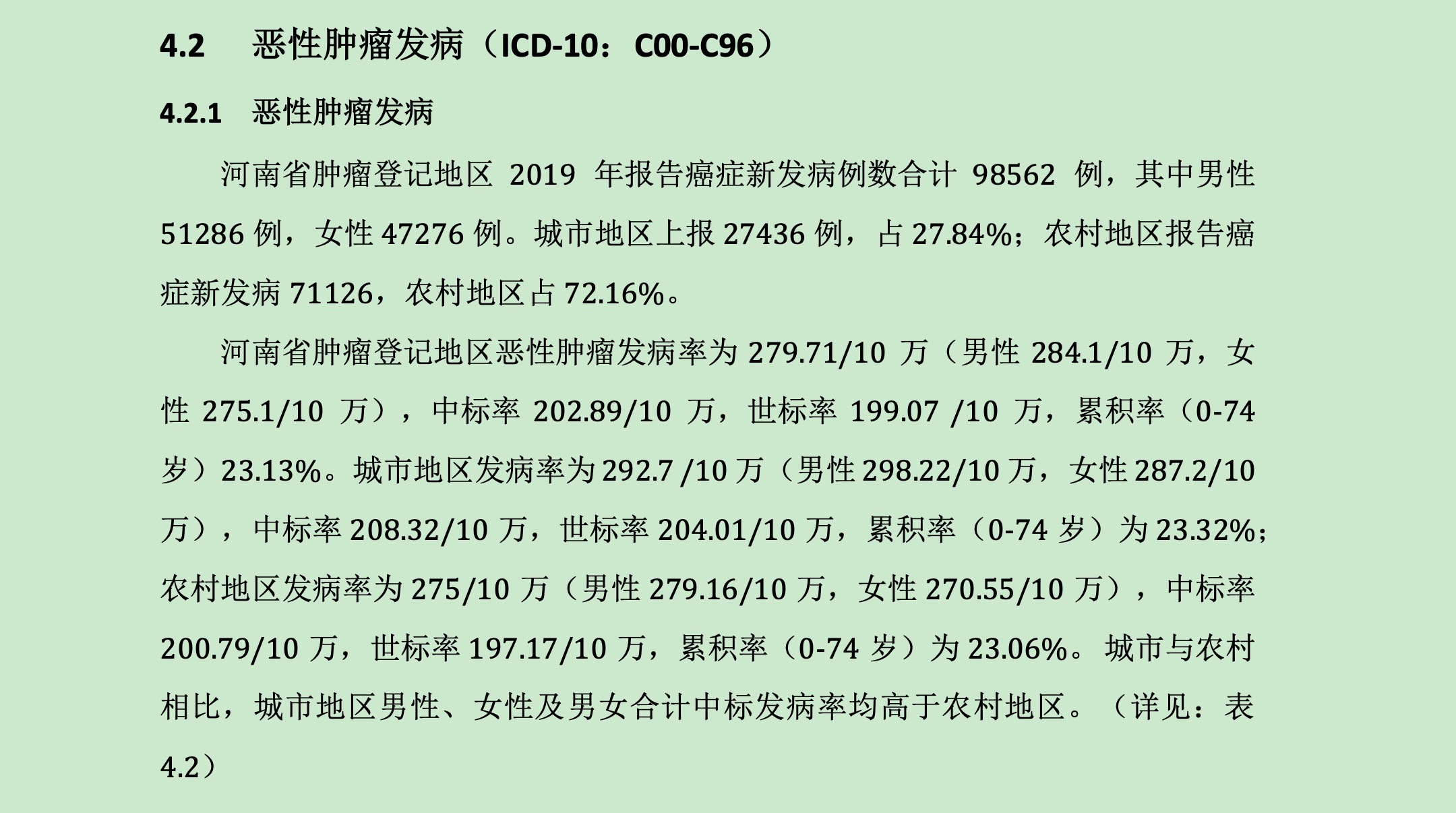
data<-inciden
header<-data.frame(
col_keys=colnames(data),
second=c("地区","性别","发病数","发病率","中标率","世标率","累积率\n0-74(%)"))
data%>%flextable()%>%
set_header_df( mapping = header, key = "col_keys" )%>%
compose(j=4,value=as_paragraph("发病率\n(1/10",as_sup("5"),")"),part="header")%>%
compose(j=5,value=as_paragraph("中标率\n(1/10",as_sup("5"),")"),part="header")%>%
compose(j=6,value=as_paragraph("世标率\n(1/10",as_sup("5"),")"),part="header")%>%
colformat_num(j=4:7,digits = 5.2)%>%
set_caption(caption= paste(year.data,"年河南省肿瘤登记地区恶性肿瘤(C00-C96)发病主要指标",""))%>%
align(align="center",part="all")%>%
bold(bold=TRUE,part="header")%>%
width(j=1:2,width=0.5)%>%
width(j=3:7,width=1.0)%>%
theme_booktabs()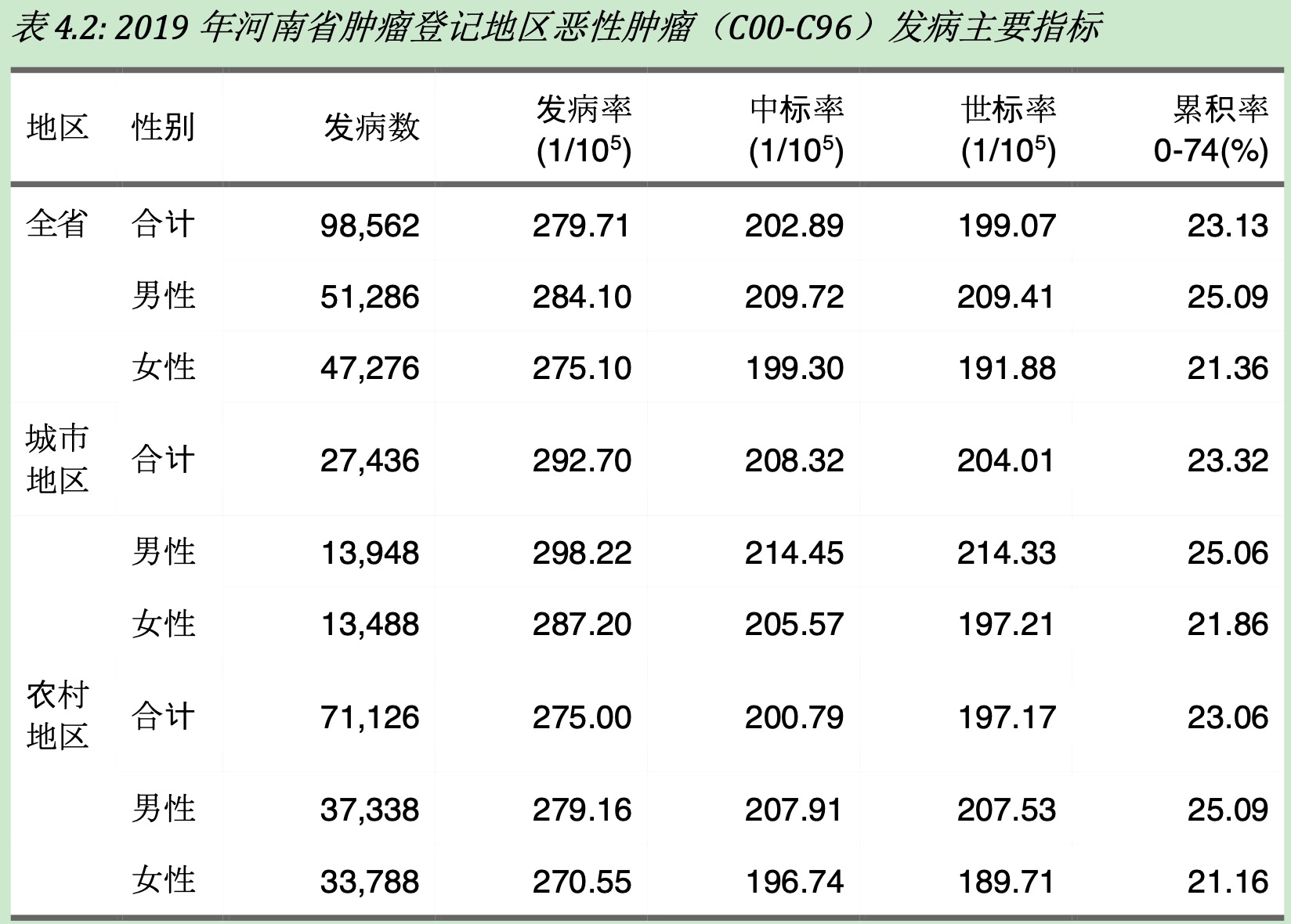
yearpub<-2023
report<-haven::read_sas("./report.sas7bdat")
areacode1 <- c("410902","411202","411303","411502","419001","411002")
areacode2 <- c("410901","411001","411201","411301","411501")
report <-report%>%
filter(city %in% c("11","2","33")|areacode %in% areacode1,
!(areacode %in% areacode2))%>%
mutate(year = as.numeric(year),
city = as.numeric(city))
source("./rscripts/gen_annual_report_plot.R")
大约需要一分钟的时间!!!
在线发布的年报更容易传播和共享
不足之处
待开发
随着肿瘤登记处数量的增加以及数据质量的提升,国家及大部分省份开始发布国家级或省级的肿瘤登记年报。
我们真的需要每年发布纸质年报吗?




R包: canregtools